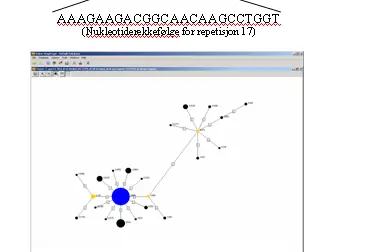
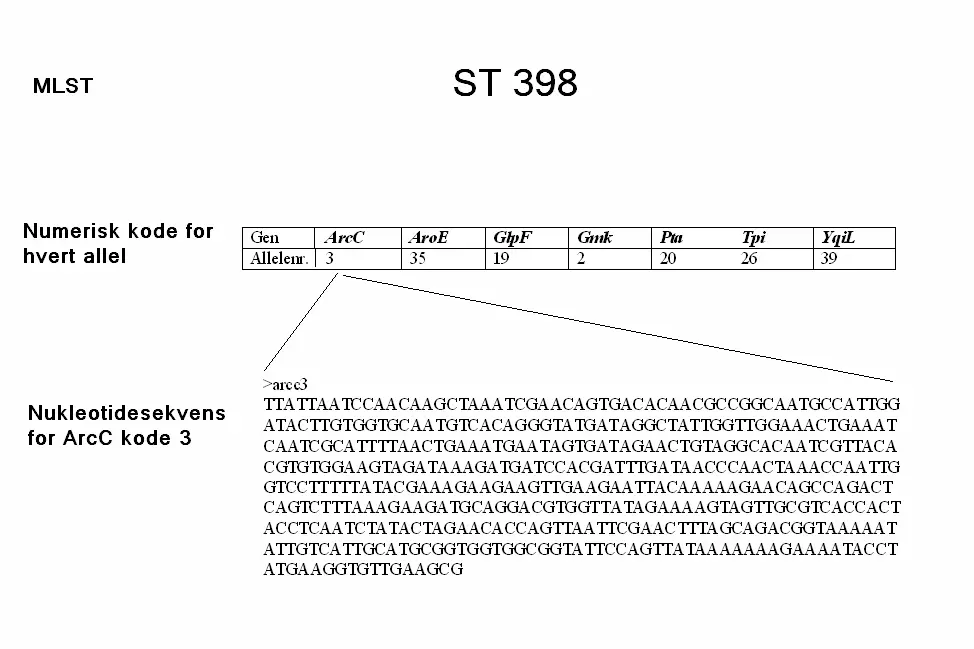
Desember 2017: Viktig informasjon fra MRSA referanselaboratorium
Oktober 2016:
Utbrudd med MRSA og Xpert MRSA NxG PCR hurtigtest
Referanselaboratoriet for MRSA har nå testet utbruddsstammen av MRSA t127 med den nye Xpert MRSA NxG testen som omfatter flere MREJ varianter enn GeneXpert MRSA testen og GeneXpert SA Nasal Complete. Her ble aktuelle genotype positiv for både mec(A/C), og SCC-mec både fra pasientmateriale og fra oppvekstkultur. Denne testen vil sånn sett kunne fange opp aktuelle utbruddsstamme i en screening. Samme forbehold som tidligere nevnt gjelder imidlertid angående PCR hurtigtester og ekstranasal screening, pooling av prøver og ved nye nye SCCmec varianter som ikke omfattes av PCR testen som benyttes.
Vær oppmerksomme på at det ikke nødvendigvis er sammenheng mellom spatype og hurtigtest. Vi har hatt andre spa t127 stammer tidligere som har testet positivt på validering av vår MRSA SA Nasal Complete test. Det er hvilken SCCmec variant spatypen har som vil være utslagsgivende for en SCCmec-orfX basert PCR deteksjon, og en spatype kan ha ulike SCC mec varianter. Om spatype t127 som har vært testet i Førde har samme SCCmec type/subtype som stammene i Tromsø vites ikke.
September 2016: Utbrudd med MRSA og negative PCR hurtigtester
I forbindelse med utbrudd av MRSA på nyfødtavdeling i Tromsø har det blitt overført to barn derfra til andre sykehus som har blitt screenet med dyrkning og PCR. Både GeneXpert SA Nasal Complete og BD Max MRSA XT har gitt negativt PCR resultat mens dyrkning har vært positiv. For GeneXpert SA Nasal Complete er aktuelle stamme også negativ for SCCmec på oppvekstkultur, mens spa genet og mecA genet var positive.
Det er velkjent at MRSA screening basert på deteksjon av orfX-SCCmec kan gi falske negative resultat sammenliknet med dyrkning. Dette skyldes at det fins mange ulike SCCmec varianter og subtyper (1). Det er altså stor genetisk variasjon i denne regionen (også kalt MREJ eller Right Extremity Junction) som medfører fare for primer mismatcher og dårligere sensitivitet ovenfor enkelte MRSA genotyper. Testenes sensitivitet vil derfor variere avhengig av lokal epidemiologi og hvilke MREJ varianter de ulike testene omfatter. Det er også kjent at mange av disse testene kun er validert og godkjent for bruk på neseprøver, og at testenes sensitivitet ofte er dårligere enn dyrkning dersom brukt på screeningprøver tatt ekstranasalt (hals, perineum). Pooling av prøvematerialer vil også kunne senke testenes sensitivitet.
Aktuelle genotype i Tromsø er spa -t127. Dette er den 5 vanligste spatypen i Norge høsten 2016. Denne genotypen er ofte assosiert med CC1 og SCCmev IV eller V. Det er tidligere beskrevet utbrudd med spa-t127 på nyfødtavdelinger både i Danmark og England (2,3). Planlagte videre analyser på referanselaboratoriet vil avdekke hvilken sekvenstype og SCCmec type utbruddsstammen i Norge tilhører.
PCR baserte MRSA hurtigtester har en høy negativ prediktiv verdi i en lavprevalenspopulasjon. MRSA referanselaboratoriet vil imidlertid oppfordre til å være varsomme med å oppheve smitteisolering basert på et negativt MRSA hurtigtestresultat. Dette gjelder særlig i høyrisikosituasjoner der det foreligger stor risiko for spredning, som på overvåknings og intensivavdelinger samt på pasienter som overføres direkte til sykehus fra land med høy forekomst av MRSA.
Ved MRSA screening skal alltid MRSA dyrkning gjøres i tillegg til eventuelle genbaserte hurtigtester for å fange opp stammer som ikke detekteres av genbaserte tester, for resistenstesting og for genotyping. Ved behov for screening av pasienter i forbindelse med aktuelle utbrudd vil nevnte to tester ikke være pålitelige. Referanselaboratoriet for MRSA vil ila. kort tid teste ut om MRSA Xpert NG fanger opp aktuelle genotype.
Referanser:
1) Hill-Cawthorne et al. Recombinations in staphylococcal cassette chromosome mec elements compromise the molecular detection of methicillin resistance in Staphylococcus aureus. PLoS One. 2014;27;9(6):e101419.
2) Ramsing BGU et al. First outbreak with MRSA in a Danish neonatal intensive care unit: risk factors and control procedures. PLoS One. 2013; 25;8(6):e66904.
3) David MD et al. Community-associated meticillin-resistant Staphylococcus aureus: nosocomial transmission in a neonatal unit. J Hosp Infect. 2006 Nov;64(3):244-50.
Desember 2015: Innsending av resistensdata
En betydelig volumøkning av innsendte MRSA stammer de siste årene gjør at MRSA referanselaboratoriet fra 2016 må prioritere sine analyser i større grad.
Vi ønsker fra 01.01.2016 å få innsendt millimetersoner for et anbefalt panel av antibiotika sammen med stammen. Genotyping vil utføres på alle stammer som tidligere, og utvalgte MRSA stammer vil fortsatt resistenstestes på nytt ved referanselaboratoriet.
Fordelen for oss blir at dette frigir kapasitet til å etablere andre nødvendige metoder (helgenomsekvensering) og metodeevalueringer. Vi ønsker samtidig å i større grad enn tidligere bruke innsendte millimetersoner til å koble ulike geno- og resistensfenotyper og synliggjøre dette i NORM rapporten. Stammer med uvanlige resistensprofiler vil retestes, eventuelt også med genotypiske metoder.
Vi har forståelse for at dette medfører noe ekstra arbeidsbelastning for hvert enkelt laboratorium. Samtidig vil vi da få frigitt kapasitet til å bruke resistensdata raskere og bedre i epidemiologisk overvåkning, se dette i relasjon til MSIS data, og gi regelmessige rapporter nasjonalt og regionalt.
For mer informasjon les vårt nyhetsbrev nr 2 som var sendt til alle laboratorier i starten av desember.
Januar 2015: LA-MRSA i Norge
De siste årene har det vært et økende fokus på dyreassosiert MRSA internasjonalt. LA MRSA tilhører CC398 og stammer opprinnelig fra MSSA CC398 hos mennesker, og har spredt seg fra mennesker til gris. Hos gris har den ervervet resistensmekasnismer mot meticillin (mecA gen) og tetracyclin (tetM gen) I tillegg har de ofte resistens mot zink (czrZ gen) som er vanlig brukt som tilsetning i dyrefor. Les videre...
Mai 2014: Månedlig rapportering
Referanselaboratoriet har opprettet en mulighet for å kunne lese månedlig rapportering av innsendte MRSA stammer fordelt på innsenderlaboratorier og på spatyper. Resultatene vil presenteres i løpet av første uke hver nye måned. Vi er takknemlige for å få tilbakemeldinger på presentasjon av dataene, særlig dersom annen eller mer utdypende informasjon ønskes. Presentasjonsformen vil evalueres på slutten av inneværende år.
Tall som fremgår i tabell 1) kan fravike fra sluttsummer og tall i øvrige tabeller da disse ekskluderer flere stammer fra samme pasient per år.
Rapporten er et øyeblikksbilde, og tenkt som en fortløpende rapportering tilbake til laboratoriene. Det er derfor ikke ønskelig at data publiseres uten nærmere henvendelse til MRSA referanselaboratoriet, slik at bedre kvalitetssikring av data er mulig.
Januar 2014: Forslag til hvilke MRSA isolat som skal testes for vankomysin heteroresistens.
Referanselaboratoriet for MRSA har i samarbeid med AFA utarbeidet et dokument og flytskjema med bakgrunnsinformasjon om vankomysinresistens hos MRSA, og om når og med hvilken metode det er mest aktuelt å undersøke for vankomysin heteroresistens (hVISA) i Norge.Dokumentet er ment som et supplement til gjeldende NordicAST dokument om påvisning av glykopeptidresistens hos MRSA. Dokumnetet har vært til høring på mikoinfo, samt hos AFA og NORM.